Zusammenspiel von HTA und Zulassung
Daten und Evidenz auf dem Weg in die klinische Forschung
Die zunehmende Verfügbarkeit von Daten und Fortschritte im Bereich der Künstlichen Intelligenz eröffnen neue Möglichkeiten für Forschung und Patientenversorgung. Real-World-Daten geben Einblicke in die realitätsnahen Auswirkungen von Therapien und können RCT ergänzen. Dafür sollten künftig Daten und Ergebnisse jeder Intervention digital verarbeitet und ausgewertet werden.
Veröffentlicht:
Obwohl molekulare und mechanistische Forschung in der Wissenschaft oft die höchste Evidenz liefern, stößt die rein auf Korrelation fokussierte evidenzbasierte Medizin mitunter an ihre Grenzen. Künstliche Intelligenz (KI) wird voraussichtlich die Verfügbarkeit und Qualität klinischer sowie anderer Real-World-Daten stark verbessern.
© Andrea Danti / stock.adobe.com
Einführung: Mit evidenzbasierter Medizin durch die Komplexität des Gesundheitswesens navigieren
Die medizinische Forschung hat ein übergeordnetes Ziel: die Gesundheit und Lebensqualität von Menschen zu verbessern. Um dies zu erreichen, werden zum einen Krankheitsmechanismen erforscht und zum anderen mögliche Interventionen entwickelt und im Anschluss auf ihre Wirksamkeit hin untersucht. Ein Eckpfeiler der medizinischen Forschung ist die evidenzbasierte Medizin (EbM), die darauf abzielt, die bestmöglichen Behandlungsentscheidungen auf Basis aktueller wissenschaftlicher Erkenntnisse zu treffen. Dieser Ansatz stützt sich auf eine Vielfalt von Forschungsmethoden, von der Grundlagenforschung im Labor bis hin zu klinischen Studien am Patienten.
In der Grundlagenwissenschaft gilt seit jeher der gemessene Wert aus Experimenten als höchste Form der Evidenz. Auch in der evidenzbasierten Medizin bildet die Grundlagenforschung das Fundament für ein tiefergehendes Verständnis der Funktionsweise potenzieller Therapien auf molekularer und zellulärer Ebene. Gemäß diesem Konzept werden grundlegende Mechanismen von Krankheiten und potenziellen therapeutischen Zielen experimentell im Labor untersucht. Die medizinische Grundlagenforschung umfasst häufig In-vitro-Studien, wie die Untersuchung der Rezeptorbindung auf molekularer Ebene, Zellkulturanalysen und präklinische In-vivo-Modelle.
Zwar stellen die Ergebnisse der Grundlagenforschung eine sehr klare Form der Evidenz dar, lassen sich aber selten direkt auf Anwendungen in lebenden Systemen übertragen, weshalb die erfolgversprechendsten Erkenntnisse weiter in der präklinischen und klinischen Forschung durch korrelative Forschung untersucht werden. Die präklinische Forschung, die den klinischen Studien vorausgeht, bildet die Brücke zwischen der Grundlagenforschung und der Anwendung beim Menschen. Ihr Fokus liegt auf der Untersuchung von Sicherheit und Wirksamkeit von Mechanismen, Medikamenten oder Behandlungen, insbesondere in Hinblick auf physiologische oder molekulare Endpunkte. Dabei werden strenge Kontrollbedingungen angewendet.
Primär nutzt die präklinische Forschung Labor- oder Tiermodelle. Doch ähnlich wie in der Grundlagenforschung ist die Übertragung präklinischer Erkenntnisse auf den Menschen ebenfalls herausfordernd, da die Ergebnisse aus Tierstudien nicht immer direkt auf den Menschen übertragbar sind. Die Extrapolation von Ergebnissen aus präklinischen Studien auf menschliche Populationen erfordert daher eine sorgfältige Berücksichtigung von physiologischen Unterschieden, genetischen Variationen und artspezifischen Reaktionen auf Therapien. Hinzukommt, dass die statistische Analyse häufig auf begrenzten Daten aus Laboruntersuchungen, Bildgebungsstudien und molekularen Essays basiert, wobei Annahmen über Normalverteilung, Homogenität der Varianz sowie Unabhängigkeit und Randomisierung getroffen werden.
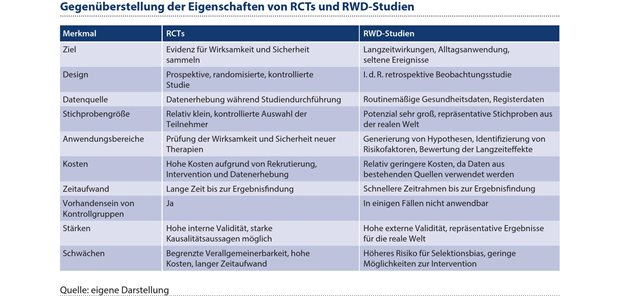
Abbildung 1: Die Tabelle zeigt einen Vergleich verschiedener Merkmale von Randomized Controlled Trials (RCTs) und Real-World-Daten-Studien.
© Springer Medizin Verlag
Im Anschluss an die Grundlagen- und präklinische Forschung bewertet die klinische Forschung die Sicherheit, Wirksamkeit und Effektivität von Behandlungen, Arzneimitteln, Diagnoseinstrumenten und Interventionen direkt in menschlichen Populationen. Im Mittelpunkt stehen patientenzentrierte Ergebnisse wie Überleben und Lebensqualität. Zu den Herausforderungen gehören ethische Erwägungen im Zusammenhang mit der Rekrutierung von Patienten, die Variabilität innerhalb menschlicher Populationen und verschiedene Störfaktoren wie Komorbiditäten und Lebensstil, da sie die Ergebnisse beeinflussen können.
Die Datenerhebung umfasst eine Vielzahl von Variablen, einschließlich standardisierter Assessmentverfahren, medizinischer Aufzeichnungen und von Patienten gemeldeter Ergebnisse. Diese Vielfalt an Messpunkten aber auch fehlende Daten, Daten-Ausreißer sowie das Vorhandensein von Störvariablen und Verzerrungen stellen häufige Herausforderungen dar, die die Datenanalyse erschweren und oft komplexe statistische Annahmen erfordern.
Der Goldstandard: Randomisierte kontrollierte Studien in der klinischen Forschung und deren Limitationen
Randomisierte kontrollierte Studien (RCTs) gelten als der Goldstandard in der klinischen Forschung, da sie die solidesten Beweise für die Wirksamkeit und Sicherheit von Interventionen liefern. Ihr Design zielt speziell darauf ab, den Einfluss der oben genannten Störfaktoren durch Randomisierung zu minimieren und eine klare Ursache-Wirkung-Beziehung zwischen der Intervention und den beobachteten Ergebnissen herzustellen.
Die Randomisierung stellt eine grundlegende Komponente von RCTs dar. Dabei werden die Teilnehmer nach dem Zufallsprinzip entweder der Interventionsgruppe oder der Kontrollgruppe zugeordnet. Die Interventionsgruppe erhält die untersuchte Behandlung, während die Kontrollgruppe ein Placebo, eine Standardbehandlung oder keine Intervention erhält. Die Randomisierung trägt dazu bei, potenzielle Störfaktoren wie zugrunde liegende Gesundheitszustände oder Lebensgewohnheiten zu kontrollieren, die die Ergebnisse beeinflussen könnten. Durch die Maskierung der Behandlungszuteilung vor den Teilnehmern und Forschern verringert eine Verblindung soweit möglich die Erkennungs- und Leistungsverzerrung.

© Cagkan / stock.adobe.com
Dieser Beitrag ist im Rahmen der Plattform zur Nutzenbewertung entstanden und im Monat Jahr in Heft 19 („Zusammenspiel von HTA und Zulassung“) erschienen.
RCTs bieten erhebliche Vorteile. Sie ermöglichen eine präzise Messung der Wirksamkeit der Behandlung und der kurzfristigen Toxizität in einem streng kontrollierten Umfeld. Dies ermöglicht eindeutige Vergleiche zwischen der Interventions- und der Kontrollgruppe und liefert einen starken Beweis für Ursache-Wirkung-Beziehungen im Gesundheitswesen.
Dennoch sind RCTs nicht uneingeschränkt für alle Situationen anwendbar. Es können sich ethische Überlegungen ergeben, die gegen die Durchführung von RCTs sprechen, insbesondere wenn der Kontrollgruppe bewährte nützliche Behandlungen vorenthalten werden. Darüber hinaus können die Rekrutierung einer ausreichenden Anzahl von Teilnehmern, die Gewährleistung einer ordnungsgemäßen Verblindung und die hohen Kosten von RCTs hohe praktische Herausforderungen darstellen.
Zudem kann das kontrollierte Umfeld von RCTs die Verallgemeinerbarkeit der Ergebnisse auf die reale klinische Praxis einschränken: Die große Variabilität der Patientenpopulationen, z. B. hinsichtlich der ethnischen Vielfalt oder des Gesundheitszustands, kann in der Regel nicht in vollem Umfang abgebildet werden. Auch die strikte Einhaltung von Behandlungsprotokollen kann die Übertragung und Anwendbarkeit in der realen Welt einschränken. Nicht zuletzt kann zum Beispiel die Untersuchung seltener Krankheiten durch RCTs aufgrund der begrenzten Anzahl verfügbarer Patienten schwierig sein, was die Gewinnung statistisch signifikanter Daten in diesem Fall schwierig gestaltet oder unter Umständen sogar unmöglich macht (Wicherski et al., 2023).
Real-World-Daten (RWD)
Aufgrund der genannten Einschränkungen von RCTs bedarf es für einen Fortschritt der klinischen Forschung dringend weiterer Herangehensweisen zur Gewinnung statistisch signifikanter Ergebnisse im klinischen Kontext. Ein Ansatzpunkt ist dabei die Analyse von Real-World-Daten (RWD). RWD umfassen alltägliche Gesundheitsdaten, die nicht für Forschungszwecke erhoben, sondern routinemäßig im Laufe der Patientenversorgung anfallen oder aus anderen Quellen stammen können. Dazu gehören beispielsweise elektronische Patientenakten, Abrechnungsdaten, Daten aus Registern oder Informationen von tragbaren Geräten in Verbindung mit Gesundheits-Apps. Diese Daten spiegeln die wahre Vielfalt der Patientenpopulation wider, inklusive Faktoren wie Alter, Geschlecht, Begleiterkrankungen und die tatsächliche Anwendung von Medikamenten in der Praxis.

Abbildung 2: Methoden basierend auf Künstlicher Intelligenz finden in zahlreichen Bereichen Anwendung. Die Abbildung zeigt ein Beispiel für den Einsatz künstlicher Intelligenz in der Wettervorhersage anhand der Methode GraphCast (Lam et al., 2023). GraphCast nutzt Mustererkennung in komplexen Wetterdaten, um präzise Vorhersagen zu erstellen. In 90 Prozent der Fälle übertraf GraphCast die Genauigkeit herkömmlicher Wettermodelle auf Supercomputernetzwerken. Der Erfolg von GraphCast zeigt das Potenzial von KI für die Analyse komplexer Systeme zur Erkennung nicht zufälliger Zusammenhänge und Muster. Es ist naheliegend, dass ähnliche Technologien in Zukunft auch in der Medizin bisherige statistische Analyseverfahren auf ähnliche Weise ergänzen oder ersetzen können. Dies kann zum Beispiel dazu beitragen, Krankheiten schneller zu identifizieren und individuelle Behandlungsstrategien in komplexen Datenfeldern auf ihre Wirksamkeit zu untersuchen.
© Springer Medizin Verlag
Die Analyse von RWD bietet einige Vorteile gegenüber RCTs (siehe Abbildung 1). Zum einen können über RWD-Studien Erkenntnisse zu Langzeitwirkungen gewonnen werden, was in RCTs aus Kostengründen häufig nicht möglich ist. Hinzukommt, dass RWD unter Real-Bedingungen erhoben wurden und Ergebnisse somit unter Umständen besser auf die praktische Anwendung beim Patienten übertragbar sind. Ein zusätzlicher Nutzen ist die Option auf Erkenntnisse zu seltenen Nebenwirkungen, die in RCTs aufgrund der kontrollierten Umgebung und begrenzten Teilnehmerzahl verborgen bleiben können. Außerdem bieten RWD insbesondere bei seltenen Erkrankungen eine Möglichkeit, überhaupt einen ausreichend großen Umfang an Daten zu erlangen.
Trotz der weitreichenden Möglichkeiten, die RWD bieten, stellt die Erhebung einer großen Menge von Daten mit dem benötigten Informationsgehalt zur Beantwortung einer spezifischen Forschungsfrage eine große Herausforderung dar. Vor allem die Beschaffung entsprechend detaillierter Informationen, wie z. B. die umfassende Erfassung von Teilnehmermerkmalen und der eindeutige Bezug von beobachteten Ergebnissen zur untersuchten Therapie, ist außerhalb der kontrollierten Umgebung von RCTs oft herausfordernd.
Zum jetzigen Zeitpunkt ist ein weiterer Nachteil von RWD oftmals die Datenqualität, die aufgrund unterschiedlicher Erfassungspraktiken in verschiedenen Einrichtungen sowie aufgrund der unterschiedlichen Zweckbestimmungen (beispielsweise Abrechnungsdaten) sehr variabel oder systematisch fehlerhaft sein kann. Daten können zudem fehlen oder inkonsistent vorliegen.
Auch wenn die Herausforderungen in der Auswertung von RWD berücksichtigt werden müssen, ist das enorme Potenzial von RWD-Studien für die klinische Forschung erheblich. Mit einer steigenden Verfügbarkeit und verbesserten Datenqualität von RWD sowie der Entwicklung neuer statistischer Analysemethoden werden RWD-Analysen zu einem immer wichtigeren Instrument auf dem Weg zu einer wirklich umfassenden Bewertung der Wirksamkeit und Sicherheit von Therapien (Wicherski et al., 2023).
Real-World-Daten für Gesundheitstechnologiebewertung nutzen
Nach heutigem Verständnis sind RCTs für die Bewertung und Zulassung von neuen Gesundheitstechnologien weiterhin zu bevorzugen. Aber es gibt bereits einige Beispiele, in denen RWD-Analysen hinzugezogen wurden. Für die Zulassung der COVID-19-Impfstoffe Comirnaty® und Vaxzevria® lieferten bestehende RCTs keine eindeutige Aussage über die Wirksamkeit bei Personen über 60 Jahren. Für eine umfassende Bewertung wurden daher RWD ausgewertet. Die RWD stammten aus elektronischen Gesundheitsakten aus Schottland, die Daten zu Hausarztversorgung, Labortests, Krankenhausaufenthalten und dem Sterberegister enthält. In Kombination mit Daten aus Impfregistern konnte hier eine prospektive Kohortenstudie in Echtzeit durchgeführt werden. Ein weiteres Beispiel ist Blinatumomab, ein Krebsmedikament. Hier ergänzte eine historische Vergleichsgruppe, basierend auf gepoolten RWD, die einarmige klinische Studie. Diese zusätzlichen Daten trugen maßgeblich zur Beschleunigung des Zulassungsverfahrens bei (Wicherski and Haenisch, 2024).
Die obigen Beispiele verdeutlichen, dass die Auswertung von RWD ein differenzierteres Bild der Wirksamkeit und Sicherheit von Gesundheitstechnologien ermöglichen und so fundiertere Entscheidungen für die Patientenversorgung getroffen werden können. Um das volle Potenzial von RWD auszuschöpfen, muss sich die Qualität dieser Daten weiter verbessern, relevante RWD-Quellen müssen leichter und umfassender zugänglich gemacht, Methoden zur Bewertung von RWD müssen weiterentwickelt und die Bedeutung von RWD in der Zulassungspraxis muss stärker etabliert werden.

Abbildung 3: Im linken Teil der Abbildung wird die bestehende, historisch gewachsene Datenumgebung des digitalen Gesundheitsökosystems eines Universitätsklinikums dargestellt. Es besteht aus vielen einzelnen Datenprozessen, die zu einem hohen Anteil noch analoge Anteile haben und in einem komplexen Geflecht nur partiell oder gar nicht interagieren.
© Springer Medizin Verlag

Im rechten Teil der Abbildung ist die Idealstruktur als Zukunftsstrategie eines solchen Systems visualisiert. Datenströme aus Versorgung, Studien und Gesundheitsdaten aus dem Alltag werden in einem patientenzentrierten Datenrückgrat interoperabel erfasst, und dem Wunsch und Erfordernissen des Patienten entsprechend für Behandlung, Forschung und Qualitätssicherung zur Verfügung gestellt.
© Springer Medizin Verlag
Künstliche Intelligenz: Unerwartetes erwarten
Unter dem Potenzial der Verfügbarkeit sehr großer Datenmengen insbesondere aus RWD kommt die Künstliche Intelligenz (KI) ins Spiel. Laut Bitkom und dem Deutschen Forschungszentrum für Künstliche Intelligenz bezeichnet KI die „Fähigkeit eines IT-Systems, ,menschliches‘ intelligentes Verhalten zu zeigen“ (Weber & Buschbacher 2017). Eine besonders spannende Entwicklung innerhalb der KI ist die generative KI. Anhand von Trainingsdaten, die zum Beispiel Bilder, Texte oder Code umfassen können, ist sie in der Lage, völlig neue Daten in komplexen Analogieschlüssen zu generieren. So kann sie beispielsweise Bilder kreieren, die einem bestimmten Stil nachempfunden sind, oder neue Musikstücke komponieren. Eine weitere wichtige Richtung der KI sind Large Language Models (LLMs). Diese basieren auf der Verarbeitung natürlicher Sprache und ermöglichen eine Kommunikation mit Maschinen in menschlicher Sprache und Denkweise. LLMs haben den Zugang zu KI-Methoden für viele Menschen und die Wissenschaft deutlich vereinfacht.
Dank LLMs können Wissenschaftler und Entwickler nun leistungsstarke KI-Tools nutzen und diese in ihre Arbeit integrieren, ohne auf teure Supercomputer angewiesen zu sein. Ein Beispiel hierfür kommt aus der Meteorologie: Mittlerweile können KI-Systeme im Laptopformat blitzschnelle Prognosen zur Wettervorhersage erstellen, die den Vorhersagen der Wetterdienste in nichts nachstehen (Voosen, 2023; siehe Abbildung 2). Ein weiteres Beispiel stammt aus der Chemie: Ein KI-Chatbot zeigte starke Leistungen bei der Vorhersage chemischer Eigenschaften und Reaktionen (Jablonka et al., 2024). Sogar Entschlüsselung historischer Dokumente profitiert von der KI, die erstmals Passagen einer zusammengerollten Papyrusrolle aus Herculaneum entschlüsselte (Marchant, 2024).
Obige Beispiele verdeutlichen, dass Multidisziplinarität für KI keine Herausforderung darstellt. Auch in der klinischen Forschung hat sie bereits Einzug gehalten. So kann KI auf allen Ebenen von klinischen Studien aber auch generell in der Vorsorge und Behandlung von Patienten einen wertvollen Beitrag zum Nutzen der Patienten leisten. Es konnte gezeigt werden, dass, im Vergleich zum manuellen Screening, KI ein vergleichbares, wenn nicht sogar besseres Screening zum Patienteneinschluss in onkologischen Studien durchführt. Dabei ist die KI aufgrund der Zeitersparnis und des verringerten Personalaufwandes dabei noch äußerst effizient (Chow et al., 2023). Ähnlich zum früher genannten Beispiel der Vorhersage chemischer Eigenschaften kann KI auch dafür genutzt werden, beispielsweise Antikörper für therapeutische Zwecke zu verbessern (Callaway, 2023).
Ein weiteres Anwendungsfeld für KI kann die Vorsorge darstellen. Nach dem Trainieren eines ChatGPT-ähnlichen Basis-Modells konnten Wissenschaftler anhand von unmarkierten Netzhautbildern Augenkrankheiten erkennen. Dieses Modell bietet die Anpassung für verschiedene Anwendungen und kann beispielsweise auch das Parkinson-Risiko evaluieren (Zhou et al., 2023). Weitere Beispiele für eine zukünftige Nutzung von KI im Umfeld von klinischen Studien gibt es in der Pharmakovigilanz (Edrees et al., 2022) sowie im Bereich der Datenkuratierung und Nutzung für die klinische Forschung (Wu et al., 2024). Um zusätzlich das Vertrauen in KI-gestützte Systeme zu erhöhen, gibt es inzwischen auch Ansätze, die KI ihre Entscheidungen nachvollziehbar erklären zu lassen, wie beispielsweise die Begründung hinter der Diagnose eines Melanomas (Chanda et al., 2024).
Ebenfalls kann KI auch Forschungseinrichtungen und klinische Studienzentren in vielerlei Hinsicht unterstützen. Das Clinical Study Center am Berlin Institute of Health at Charité (BIH) arbeitet an KI-Anwendungen, um beispielsweise die Entwicklung von standardisierten Studienprotokollen zu unterstützen, die Datenqualität zu verbessern und Patienten im Rahmen eines erweiterten Einverständnisprozesses (Broad Consent) umfassender zu informieren und in ihre Behandlungsentscheidungen einzubinden.
Datenqualität und Datennutzung
Hochwertige Daten und deren effektive Nutzung sind der Schlüssel für eine erfolgreiche medizinische Forschung. Um diese Ziele zu erreichen, ist die enge Zusammenarbeit zwischen Forschern, Patienten und ihren Vertretern unerlässlich. Grundlage dafür ist die patientenorientierte klinische Forschung, bei der der Patient im Mittelpunkt steht, um unter Einbeziehung der Betroffenen die bestmögliche Behandlung für jeden einzelnen Patienten zu finden.
Patientenorganisationen und Interessenvertretungen können dabei klinische Studien aktiv mitgestalten. Ziel der patientenorientierten klinische Forschung ist weiterhin die Entwicklung von Präventionsstrategien, die Verbesserung der Behandlung bei hoher Lebensqualität von Patienten sowie die Optimierung von Strukturen im Gesundheitssystem und die Erstellung von für Patienten verständlichen Informationen und Studienergebnissen.
Zur Erreichung der oben geschilderten Ziele der patientenorientierten Forschung ist die Analyse von RWD, also der Analyse großer und komplexer Datensätze unabdingbar und bietet vielfältige Möglichkeiten. Noch ist der Zugang zu solchen Daten aus verschiedenen Gründen erschwert. Ein Grund dafür ist zum Beispiel, dass unser digitales Gesundheitsökosystem derzeit ein komplexes System ist, welches aus vielen isolierten Datenverarbeitungs- und -speicherprozessen besteht (siehe Abbildung 3, links). Klinische Studien stellen dabei eine Ausnahme dar, da hier Daten in der Regel zentral erfasst werden.
Mit den verschiedenen neuen Digitalgesetzen wie beispielsweise dem Gesundheitsdatennutzungsgesetz soll der Datenaustausch in Zukunft vorangetrieben werden. Ein systematischer Datenaustausch legt den Grundstein, um die aktuellen Innovationen sowohl in der Medizin als auch in den Methodiken der Datenanalyse zu ermöglichen bzw. zu fördern und so tatsächlich einen Mehrwert für die Patienten zu generieren. Ziel muss es sein, eine interoperable Umgebung zu schaffen, in der Daten problemlos aus verschiedenen Projekten auch sektorenübergreifend ausgetauscht und gemeinsam ausgewertet werden können (siehe Abbildung 3, rechts).
Die aktuellen Entwicklungen in allen beteiligten Bereichen bergen großes Potenzial. So könnte künftig für jeden Patienten quasi eine maßgeschneiderte klinische Studie durchgeführt werden. Dabei ist zu beachten, dass sich ein erheblicher Anteil der Patienten mit seltenen Erkrankungen in der Regelversorgung befinden, ohne dass ihre Krankheit diagnostiziert wird. Durch die intelligente Verknüpfung von Daten aus verschiedenen Quellen könnten solche Erkrankungen künftig schneller erkannt und zielgerichtete Therapien entwickelt werden.
Von der Forschung zum Patienten und zurück: Die Kraft der Translation
Um erzielte Fortschritte aus der Forschung schnell zu den Patienten zu bringen braucht es die Translation – die Übertragung von Erkenntnissen aus der Grundlagenforschung in die klinische Anwendung und umgekehrt. Um die Translation zum Nutzen des Patienten noch weiter voranzubringen, brauchen wir ein Umdenken. In der Onkologie brauchen wir eine Vision Zero.
Vision Zero ist ein ursprünglich aus der Verkehrssicherheitsforschung stammendes Konzept. Ziel der Vision Zero ist die Verhinderung aller vermeidbaren Todesfälle in dem jeweiligen Bereich. Fokus früherer Strategien in der Unfallforschung war häufig die Senkung der Gesamtzahl von Kollisionen. Als Hauptunfallursache wurden menschliche Fehler ausgemacht. Die Folge waren kostspielige Maßnahmen, die das menschliche Verhalten auf einem unvollkommenen Straßensystem verbessern sollten.
Ein neuer Ansatz der Translation ermöglichte ein Umdenken und brachte neue Erkenntnisse hervor. Anstatt nur die Anzahl der Unfälle zu minimieren, lag der Fokus nun auf der Verhinderung von Todesfällen und schweren Verletzungen. Dabei wurden Mängel im Verkehrssystem selbst als Unfallursache identifiziert und der Fokus lag darauf, durch perfekte Straßensysteme das imperfekte menschliche Verhalten auszugleichen.
Vision Zero in der Onkologie bedeutet, Krebsentstehung wo möglich zu verhindern und die Heilungschance für alle Krebserkrankungen deutlich zu verbessern oder zumindest Patienten mit Krebs in Zukunft eine durchgehend hohe Lebensqualität zu ermöglichen. Zugrundeliegende Konzepte umfassen Präventionsmaßnahmen, Früherkennung, verbesserte Behandlungsmethoden und eine umfassende Versorgung. Es geht also darum, die gesamte Palette der Möglichkeiten auszuschöpfen, also jeden Stein umzudrehen, um Leben zu retten und das Leiden durch Krebs zu minimieren.
Ein Beispiel für gelungene Translationsforschung aus dem Bereich Onkologie ist die Umsetzung nationaler Programme für genomische Medizin. Das Deutsche Konsortium für erblichen Brust- und Eierstockkrebs (DKH-finanziert) und das nationale Netzwerk Genomische Medizin (nNGM) Lungenkrebs (DKH-finanziert) erforschen genetische Zusammenhänge bei Krebserkrankungen, und schließt mittlerweile fast zwei Drittel der betroffenen Patienten in Deutschland. Am Beispiel des nNGMProgramms zeigt sich, dass involvierte Patienten in der Versorgung nachweislich von einer signifikant höheren Überlebensrate profitieren (Kästner et al., 2024).
Fazit: Medizin der Zukunft – Daten, KI und neue Maßstäbe
Obwohl molekulare und mechanistische Forschung in der Wissenschaft oft die höchste Evidenz liefern, stößt die rein auf Korrelation fokussierte evidenzbasierte Medizin mitunter an ihre Grenzen. Künstliche Intelligenz (KI) wird voraussichtlich die Verfügbarkeit und Qualität klinischer sowie anderer Real-World-Daten stark verbessern. KI-gestützte Analysen und moderne statistische Verfahren haben das Potenzial, auf Basis dieser Daten randomisierte klinische Studien zu ergänzen oder – bei hoher Qualität der Beobachtungsdaten und prospektivem Design – sogar zu ersetzen.
Daher sollten zukünftige die Daten und Ergebnisse jeder medizinischen Intervention digital verarbeitet und ausgewertet werden, so wie es in anderen Lebensbereichen für sicherheitsrelevante oder sogar überlebenswichtige Technologien und Maßnahmen selbstverständlich ist. Dann kann das enorme Potenzial einer umfassenden Datenbasis ausgeschöpft werden, um noch zielgerichtetere und effektivere Therapie- und Diagnoseverfahren zu entwickeln und die medizinische Versorgung zum Wohle der Patienten weiter entscheidend zu verbessern.

© Simone Baar
Prof. Dr. Christof von Kalle
ist seit Juni 2019 BIH-Chair für Klinisch-Translationale Wissenschaften und Gründungsdirektor des gemeinsamen Clinical Study Centers des Berlin Institute of Health at Charité (BIH) und Charité – Universitätsmedizin Berlin. Der Wissenschaftler mit klinischem Hintergrund in der Hämatologie/Onkologie ist seit 2024 Sachverständiger im Aufsichtsrat des Universitätsklinikums Freiburg und war von 2019 bis 2023 Mitglied im Sachverständigenrat zur Begutachtung der Entwicklung im Gesundheitswesen. Zuvor war er von 2005 bis 2018 Gründungsdirektor des Nationalen Centrums für Tumorerkrankungen (NCT) Heidelberg.

© Simone Baar
Dr. Stefanie Rudolph
ist Biologin und arbeitet seit 2019 als wissenschaftliche Referentin am Clinical Study Center des Berlin Institute of Health (BIH) at Charité insbesondere mit dem Fokus auf Digitalisierungsprozesse im onkologischen Kontext. Zuvor war sie am NCT Heidelberg tätig.

© Privat
Dr. Mona Rams
studierte Bioinformatik und promovierte in diesem Fachbereich mit einem Schwerpunkt auf maschinellem Lernen und Algorithmenentwicklung für die Präzisionsonkologie. Seit 2023 ist sie wissenschaftliche Referentin am Clinical Study Center des Berlin Institute of Health (BIH) at Charité.
Literatur
1 Callaway E. 2023. How generative AI is building better antibodies. Nature 617:235.
2Chanda T, Hauser K, Hobelsberger S, Bucher T-C, Garcia CN, Wies C, Kittler H, Tschandl P, Navarrete-Dechent C, Podlipnik S, Chousakos E, Crnaric I, Majstorovic J, Alhajwan L, Foreman T, Peternel S, Sarap S, Özdemir İ, Barnhill RL, Llamas-Velasco M, Poch G, Korsing S, Sondermann W, Gellrich FF, Heppt MV, Erdmann M, Haferkamp S, Drexler K, Goebeler M, Schilling B, Utikal JS, Ghoreschi K, Fröhling S, Krieghoff-Henning E, Salava A, Thiem A, Dimitrios A, Ammar AM, Vučemilović AS, Yoshimura AM, Ilieva A, Gesierich A, Reimer-Taschenbrecker A, Kolios AGA, Kalva A, Ferhatosmanoğlu A, Beyens A, Pföhler C, Erdil DI, Jovanovic D, Racz E, Bechara FG, Vaccaro F, Dimitriou F, Rasulova G, Cenk H, Yanatma I, Kolm I, Hoorens I, Sheshova IP, Jocic I, Knuever J, Fleißner J, Thamm JR, Dahlberg J, Lluch-Galcerá JJ, Figueroa JSA, Holzgruber J, Welzel J, Damevska K, Mayer KE, Maul LV, Garzona-Navas L, Bley LI, Schmitt L, Reipen L, Shafik L, Petrovska L, Golle L, Jopen L, Gogilidze M, Burg MR, Morales-Sánchez MA, Sławińska M, Mengoni M, Dragolov M, Iglesias-Pena N, Booken N, Enechukwu NA, Persa O-D, Oninla OA, Theofilogiannakou P, Kage P, Neto RRO, Peralta R, Afiouni R, Schuh S, Schnabl-Scheu S, Vural S, Hudson S, Saa SR, Hartmann S, Damevska S, Finck S, Braun SA, Hartmann T, Welponer T, Sotirovski T, Bondare-Ansberga V, Ahlgrimm-Siess V, Frings VG, Simeonovski V, Zafirovik Z, Maul J-T, Lehr S, Wobser M, Debus D, Riad H, Pereira MP, Lengyel Z, Balcere A, Tsakiri A, Braun RP, Brinker TJ, Reader Study C. 2024. Dermatologist-like explainable AI enhances trust and confidence in diagnosing melanoma. Nature Communications 15:524. doi: 10.1038/s41467-023-43095-4. https://doi.org/10.1038/s41467-023-43095-4
3 Chow R, Midroni J, Kaur J, Boldt G, Liu G, Eng L, Liu F-F, Haibe-Kains B, Lock M, Raman S. 2023. Use of artificial intelligence for cancer clinical trial enrollment: a systematic review and meta-analysis. JNCI: Journal of the National Cancer Institute 115:365-374. doi: 10.1093/jnci/djad013. https://doi.org/10.1093/jnci/djad013
4 Edrees H, Song W, Syrowatka A, Simona A, Amato MG, Bates DW. 2022. Intelligent Telehealth in Pharmacovigilance: A Future Perspective. Drug Safety 45:449-458. doi: 10.1007/s40264-022-01172-5. https://doi.org/10.1007/s40264-022-01172-5
5 Jablonka KM, Schwaller P, Ortega-Guerrero A, Smit B. 2024. Leveraging large language models for predictive chemistry. Nature Machine Intelligence 6:161-169. doi: 10.1038/s42256-023-00788-1. https://doi.org/10.1038/s42256-023-00788-1
6 Kästner A, Kron A, van den Berg N, Moon K, Scheffler M, Schillinger G, Pelusi N, Hartmann N, Rieke DT, Stephan-Falkenau S. 2024. Evaluation of the effectiveness of a nationwide precision medicine program for patients with advanced non-small cell lung cancer in Germany: a historical cohort analysis. The Lancet Regional Health–Europe 36. doi: 10.1016/j.lanepe.2023.100788
7 Marchant J. 2024. First passages of rolled-up Herculaneum scroll revealed. Nature 626:461-462.
8 Lam R, Sanchez-Gonzalez A, Willson M, Wirnsberger P, Fortunato M, Alet F, Ravuri S, Ewalds T, Eaton-Rosen Z, Hu W, Merose A, Hoyer S, Holland G, Vinyals O, Stott J, Pritzel A, Mohamed S, Battaglia P. 2023. ‚Learning skillful medium-range global weather forecasting‘, Science, 382: 1416-21. https://doi.org/10.1126/science.adi2336
9 Voosen P. 2023. AI is set to revolutionize weather forecasts. Science (New York, NY) 382:748-749.
10 Weber M, Buschbacher F. 2017. ‚Künstliche Intelligenz–Wirtschaftliche Bedeutung, gesellschaftliche Herausforderungen, menschliche Verantwortung‘, Bitkom e. V., DFKI, Berlin, Kaiserslautern.
11 Wicherski J, Haenisch B. 2024. Die Anwendung von Real-world-Evidenz in Entscheidungsprozessen der Arzneimittelregulation. Bundesgesundheitsblatt - Gesundheitsforschung - Gesundheitsschutz 67:149-154. doi: 10.1007/s00103-023-03830-0. https://doi.org/10.1007/s00103-023-03830-0
12 Wicherski J, Schneider K, Zinserling J, Heß S, Haenisch B, Broich K. 2023. Real-world-Daten in der Arzneimittelregulation – aktuelle Entwicklungen und Ausblick‘, Prävention und Gesundheitsförderung. https://doi.org/10.1007/s11553-022-01010-7
13 Wu X, Li W, Tu H. 2024. Big data and artificial intelligence in cancer research. Trends in Cancer 10:147-160. https://doi.org/10.1016/j.trecan.2023.10.006
14 Zhou Y, Chia MA, Wagner SK, Ayhan MS, Williamson DJ, Struyven RR, Liu T, Xu M, Lozano MG, Woodward-Court P, Kihara Y, Allen N, Gallacher JEJ, Littlejohns T, Aslam T, Bishop P, Black G, Sergouniotis P, Atan D, Dick AD, Williams C, Barman S, Barrett JH, Mackie S, Braithwaite T, Carare RO, Ennis S, Gibson J, Lotery AJ, Self J, Chakravarthy U, Hogg RE, Paterson E, Woodside J, Peto T, McKay G, McGuinness B, Foster PJ, Balaskas K, Khawaja AP, Pontikos N, Rahi JS, Lascaratos G, Patel PJ, Chan M, Chua SYL, Day A, Desai P, Egan C, Fruttiger M, Garway-Heath DF, Hardcastle A, Khaw SPT, Moore T, Sivaprasad S, Strouthidis N, Thomas D, Tufail A, Viswanathan AC, Dhillon B, Macgillivray T, Sudlow C, Vitart V, Doney A, Trucco E, Guggeinheim JA, Morgan JE, Hammond CJ, Williams K, Hysi P, Harding SP, Zheng Y, Luben R, Luthert P, Sun Z, McKibbin M, O’Sullivan E, Oram R, Weedon M, Owen CG, Rudnicka AR, Sattar N, Steel D, Stratton I, Tapp R, Yates MM, Petzold A, Madhusudhan S, Altmann A, Lee AY, Topol EJ, Denniston AK, Alexander DC, Keane PA, Eye UKB, Vision C. 2023. A foundation model for generalizable disease detection from retinal images. Nature 622:156-163. doi: 10.1038/s41586-023-06555-x. https://doi.org/10.1038/s41586-023-06555-x